= déterminer l'ordre d'enchaînement des nucléotides pour un fragment d'ADN donné
Technique de Sanger
= déterminer l'ordre d'enchaînement des nucléotides pour un fragment d'ADN donné
Technique de Sanger
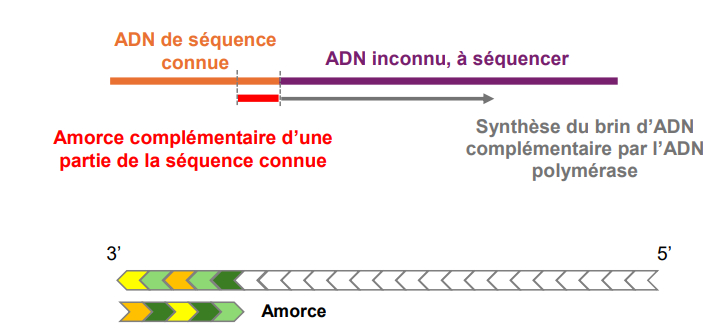
on chercher à connaître les séquences en blanc
Principe :
-> principe du séquençage
-> pour satisfaire aux points 1 et 3 il faut :
solution : réaliser la réaction en présence d'un didésoxyribonucléotide
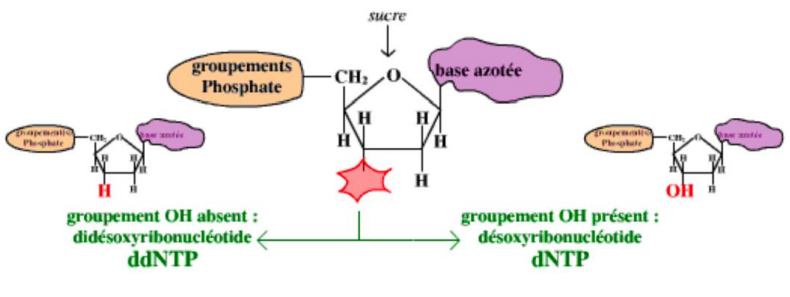
didésoxyribonucléotide
-> groupement OH absent contrairement à un désoxyribonucléotide classique
-> lorsqu'une ADN polymérase utilise un ddNTP au lieu d'un dNTP, elle n'est plus capable de rajouter le moindre nucléotide à la suite = fin de la séquence d'ADN
-> si elle ajoute un dNTP, elle ajoute des nucléotides à la suite = synthèse du brin se poursuit
-> répartition de l'ADN à séquencer en 4 tubes
-> en commun dans les 4 lots :
-> différences des tubes : ddNTP spécifiques
-> ADN polymérase va :
= choix aléatoire par la polymérase
= localisation aléatoire du site de blocage
= obtention de tous les brins possibles
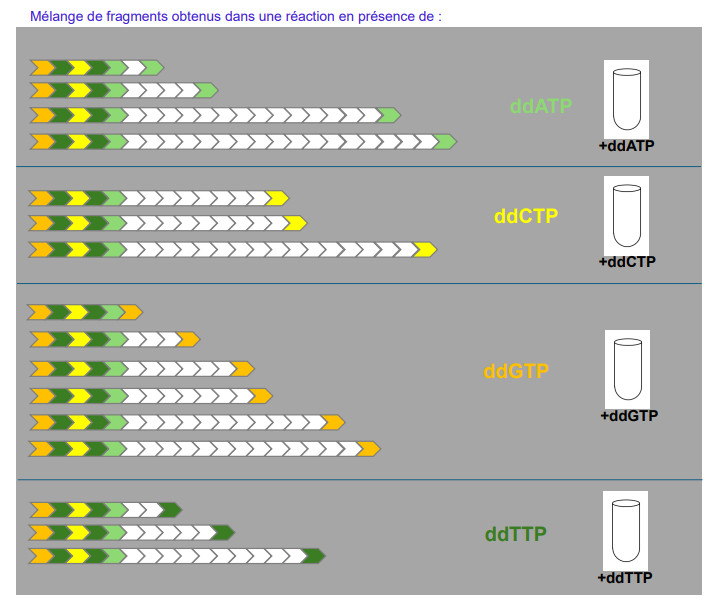
Mélange de fragments obtenus dans une réaction en présence de :
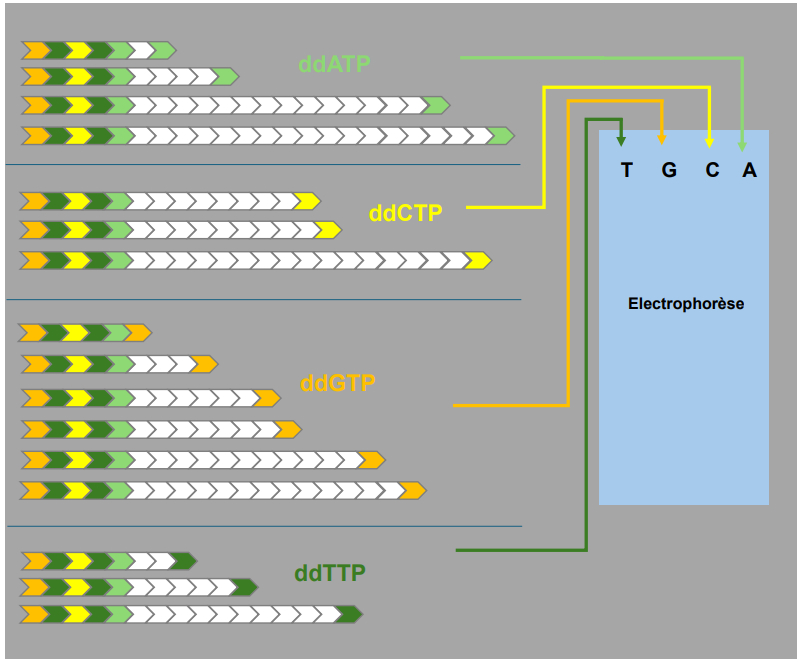
Chargement du mélange de chaque réaction dans un puit d'un gel de polyacrylamide
piste A -> tous les fragments d'ADN néo synthétisés se terminent par un "A"
piste T -> tous les fragments d'ADN néo synthétisés se terminent par un "T"
etc...
= sépare les fragments en fonction de leur taille -> le plus petit sera tout en bas - plus le fragment est petit + il migre loin du puits
-> réaction

-> on réalise la séquence complémentaire et antiparallèle pour obtenir la séquence en blanc
Remarques :
= déterminer l'ordre d'enchaînement des nucléotides pour un fragment d'ADN donné
Technique de Sanger

on chercher à connaître les séquences en blanc
Principe :
-> principe du séquençage
-> pour satisfaire aux points 1 et 3 il faut :
solution : réaliser la réaction en présence d'un didésoxyribonucléotide

didésoxyribonucléotide
-> groupement OH absent contrairement à un désoxyribonucléotide classique
-> lorsqu'une ADN polymérase utilise un ddNTP au lieu d'un dNTP, elle n'est plus capable de rajouter le moindre nucléotide à la suite = fin de la séquence d'ADN
-> si elle ajoute un dNTP, elle ajoute des nucléotides à la suite = synthèse du brin se poursuit
-> répartition de l'ADN à séquencer en 4 tubes
-> en commun dans les 4 lots :
-> différences des tubes : ddNTP spécifiques
-> ADN polymérase va :
= choix aléatoire par la polymérase
= localisation aléatoire du site de blocage
= obtention de tous les brins possibles

Mélange de fragments obtenus dans une réaction en présence de :

Chargement du mélange de chaque réaction dans un puit d'un gel de polyacrylamide
piste A -> tous les fragments d'ADN néo synthétisés se terminent par un "A"
piste T -> tous les fragments d'ADN néo synthétisés se terminent par un "T"
etc...
= sépare les fragments en fonction de leur taille -> le plus petit sera tout en bas - plus le fragment est petit + il migre loin du puits
-> réaction

-> on réalise la séquence complémentaire et antiparallèle pour obtenir la séquence en blanc
Remarques :