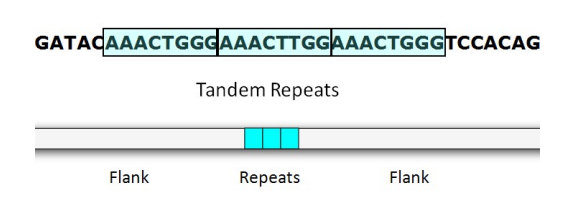
Séquences répétées en tandem
= séquences plus ou moins longues appelées "unités de répétition" (ex : AAACTGGG) et se répètent successivement un certain nombre de fois
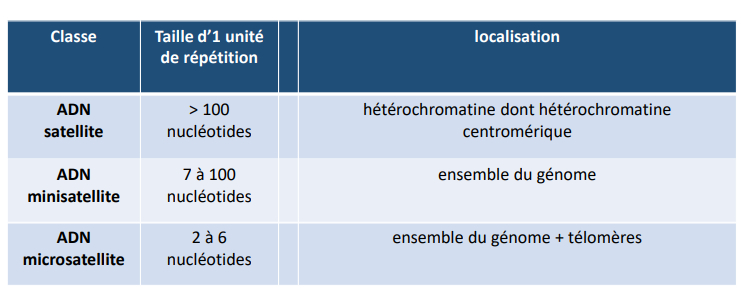
-> plusieurs classes en fonction de la longueur des unités de répétition
- ADN satellite
- ADN minisatellite
- ADN microsatellite
-> variation du nombre de répétitions d'un individu à un autre
-> rôle des minisatellites localisés au niveau des région régulatrices et au niveau du promoteur des gènes
- impliqués dans la régulation de l'expression génique en servant de site de fixation pour les facteurs de transcription = augmentation du nombre de répétition des motifs = augmentation du nombre de site de fixation pour les facteurs de transcription
-> rôle des microsatellites localisés au niveau des régions régulatrices et au niveau du promoteur des gènes
- même rôle que les minisatellites
-> ex de maladie liée au microsatellites :
- maladie de Huntignton due à une anomalie du nombre de répétitions d'un triple nucléotide (CAG) sur un gène de Huntingtine
- HTT mutée = agrégation et formation de fragments toxiques = mort cellulaire
- maladie de l'X fragime = 1ère cause de retard mental héréditaire
- déficit intellectuel, troubles du comportement et anomalies physiques
- liée à une répétition anormale d'un triplet de nucléotides (CGG)
- chez les garçons : retard mental, troubles du comportement, du langage, visage allongé
- chez les filles : tableau clinique + modéré, timidité et anxiété sociale
Eléments transposables
= séquence d'ADN mobiles capables de se déplacer ou de se copier d'un endroit à un autre sur les chromosomes
-> 4 classes :
- transposons à ADN
- rétrotransposons sans LTR
- rétrotransposons à LTR (long terminal repeat)
- rétrovirus endogènes
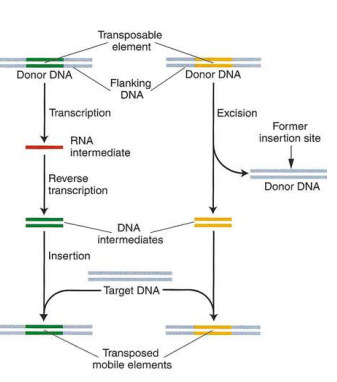
-> modes de transpositions
- transposition conservatrice (couper-coller) : par migration directe, excision de l'élément transposable et insertion dans une nouvelle localisation = transposons à ADN
- transposition réplicative (copier-coller) : duplication et insertion dans une nouvelle localisation = rétrotransposons sans LTR, rétrotransposons à LTR, rétrovirus endogènes
Rétrotransposons sans LTR
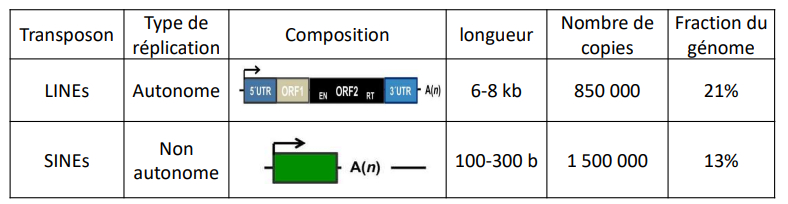
LINEs = longues séquences répétées dispersées au sein du génome
- 3 familles : LINE-1, LINE-2, LINE-3
- protéine ORF1p possède une activité chaperon des acides nucléiques et ORF2p possède des activités endonucléase et de transcriptase inverse
SINEs = courtes séquences répétées dispersées au sein du génome
- utilisation de la machinerie des LINEs (transposons non autonomes)
- insertion impliquées dans plusieurs maladies humaines
-> impliqués dans la régulation de l'expression génique en servant de site de fixation à des facteurs de transcription
-> conséquences de l'insertion de LINE-1 au niveau d'une séquence régulatrice d'un gène = modification de l'expression de ce gène
-> conséquences possibles de l'insertion de LINE-1 au sein d'un exon = mutagenèse insertionnelle pouvant notamment conduire à l'inactivation d'un gène
-> conséquences possibles de l'insertion de LINE-1 au sein de l'intron = altération du processus d'épissage
Exemple des cellules cancéreuses :
- forte méthylation des dinucléotides CpG au niveau des éléments transposables de type LINEs dans les cellules normales -> répression de l'expression de ces rétrotransporteurs sans LTR
- hypométhylation des dinucléotides CpG au niveau des éléments transposables de type LINE-1 dans les cellules cancéreuses -> transcription suivie de l'insertion de nouvelles copies dans le génome : conséquences = augmentation de l'expression de gènes codant pour des protéines favorisant la progression des cancers ET inhibition de l'expression ou "inactivation" des gènes codant des protéines exerçant une activité anti-tumorale
-> séquences LINE ne se déplacent pas dans les cellules normales uniquement dans les cellules cancéreuses
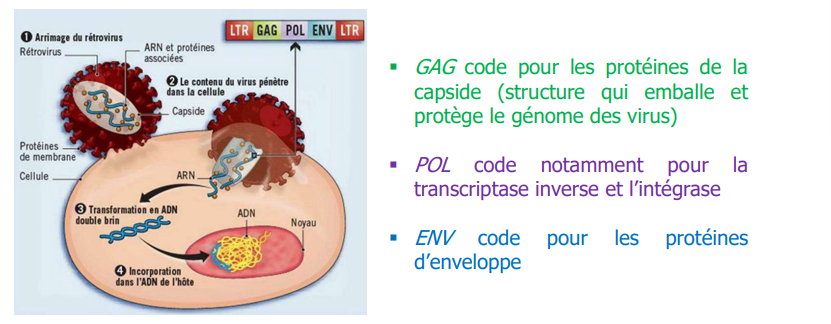
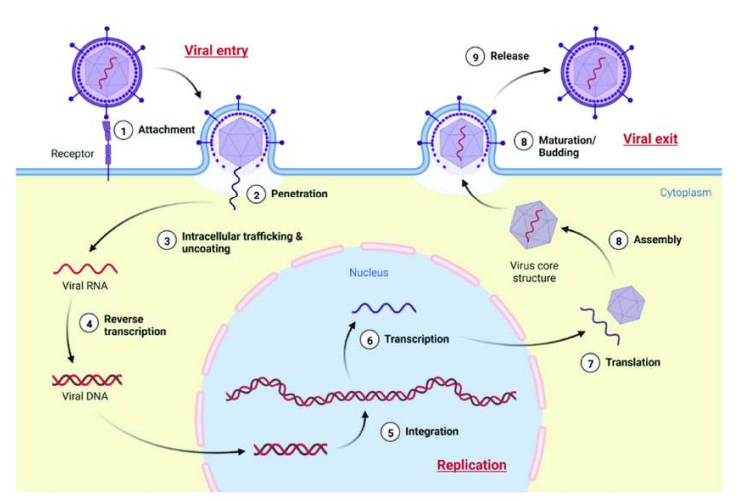
Rétrovirus endogènes : séquences de rétrovirus faisant partie intégrante du génome de l'organisme où on l'a identifié
-> structure comparable à celle des rétrovirus "classiques"
-> enveloppe sert à fusionner avec une cellule cible
-> capable de rentrer dans une cellule = perte de l'enveloppe et de la caspide virale
- ADN se retrouve dans le cytoplasme de la cellule hôte
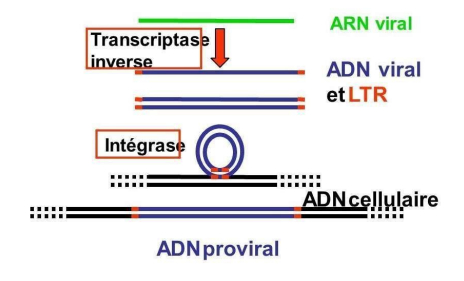
- transcriptase inverse va convertir l'ARN viral en ADN viral
- cet ADN entre dans le noyau et grâce à l'intégrase et aux séquences LTR va pouvoir rentrer dans le génome de la cellule hôte donc l'ADN cellulaire va être transcrit grâce à la machinerie de la cellule hôte
- transcription permet la synthèse des capsides, enveloppes...= production de virions
-> au cours de l'évolution la majorité des rétrovirus endogènes ont été inactivé
-> exemple de la syncytine codée par des rétrovirus endogènes dans la formation du syncytiotrophoblaste
- certains rétrovirus restent capables de produire des protéine
- infection d'une espèce ancestrale -> intégration du rétrovirus dans les cellules germinales et transmission à la descendance = inactivation du rétrovirus et conservation de la séquence codant l'enveloppe virale = gène de la syncytine
A savoir +++
-> certaines séquences répétitives ont la faculté de se déplacer dans le génome
-> les rétrotransposons sont des ADN qui transcrivent des ARN
-> les séquences LINEs et SINEs utilisent le mode de transposition réplicative
-> les 4 classes
-> les modes de transpositions
-> l'utilité du site de fixation